《自然-方法》新闻截图
南开新闻网讯 近日,《自然-方法》杂志刊发题为“Critical assessment of protein intrinsic disorder prediction”的研究论文并配发题为“A community effort to bring structure to disorder”的新闻,报道了国际蛋白质无序区域预测比赛(CAID)的详情。我校统计与数据科学学院教授胡刚、数学科学学院副教授王奎与美国弗吉尼亚联邦大学教授Lukasz Kurgan等合作开发的预测方法fIDPnn在此项比赛中表现优异,多项指标取得第一名。
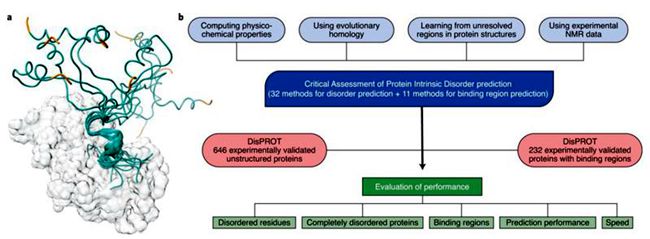

蛋白质无序区域预测比赛流程和结果
一般认为,蛋白质折叠为固定的结构来行使功能,而近年来的研究表明,蛋白质中存在一些并没有固定结构的“无序区域”,这些区域在蛋白质与其他分子相互作用中起着关键作用,参与了信号传导、转录、翻译等重要的细胞功能。蛋白质中存在大量这样的无序区域,例如人类蛋白质中的30%-40%被认为是无序的。而由于实验测定蛋白质结构的困难,计算方法对这些无序区域的预测是研究这些无序区域的主要途径之一。本次比赛则是针对蛋白质无序区域预测的首次国际比赛,吸引了国际上从事蛋白质无序区域研究的数十个团队参加。
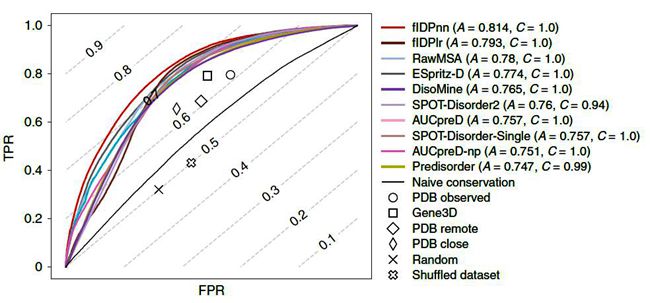

fIDPnn与其他方法的性能指标比较
本次比赛对参赛的数十种方法进行了详细评估。胡刚教授等开发的方法fIDPnn使用机器学习的方法,结合蛋白质序列的多种特征对蛋白质无序区域进行了准确预测,在Disprot标准、Disport-PDB标准和全无序序列预测三项评比中取得两项第一名(Disport标准和全无序序列预测),在Disport-PDB标准下也名列前茅。预测方法fIDPnn不仅预测准确率领先,而且运行速度较之竞争对手快了10倍以上,极大方便了蛋白质无序区域的研究,也给进一步研究这些区域的功能提供了重要参考。
胡刚,统计与数据科学学院教授,博士生导师,长期从事结构生物信息学、单细胞多组学分析等方向的研究工作,以主要作者在Circulation, Nature Communications, Bioinformatics等权威杂志发表学术论文多篇。
王奎,数学科学学院副教授,硕士生导师,主要从事结构生物信息学、单细胞RNA测序分析,深度学习在生物信息中的应用等方向的研究工作,以主要作者在Acta Neuropathol,Bioinformatics,Structure等杂志发表论文多篇。(统计与数据科学学院供稿)
比赛详情请参见:
https://www.nature.com/articles/s41592-021-01123-5
https://www.nature.com/articles/s41592-021-01117-3