2024年11月30日,第16届世界蛋白质结构预测比赛(CASP16)结果正式公布,南开大学统计与数据科学学院郑伟教授带领的团队在蛋白质核酸复合物结构预测、复合物整体折叠精度估计、蛋白质多构象结构预测等多个赛道获得第一名,在核酸多聚体结构预测中服务器组位居第一,并受邀在CASP16赛后,多米尼加共和国举办的国际会议上作特邀报告。

Critical Assessment of Structure Prediction (CASP)是国际著名的蛋白质结构预测比赛,被誉为蛋白质结构预测领域的奥林匹克竞赛。自1994年由马里兰大学的John Moult教授等人创办以来,CASP每两年举办一次,旨在对大分子和复合物结构建模的计算方法进行严格评估,推动领域内技术进步。
第16届CASP比赛于2024年5月至8月举行,历时四个月。期间,CASP组织者发布尚未公开的大分子和复合物实验结构,参赛团队需要在限定时间内提交预测的结构模型。赛事涵盖了多个赛道,包括蛋白质单体结构预测、蛋白质复合物结构预测、准确性评估、核酸结构预测、配体复合物结构预测以及大分子多构象预测等。本届赛事吸引了来自全球209个团队的参与,包括南开大学、山东大学、华中科技大学、中国科学技术大学、复旦大学、清华大学、中山大学、密歇根大学、密歇根州立大学、密苏里大学、普渡大学、斯德哥尔摩大学、新加坡国立大学等众多知名高校与晶泰科技等业界知名研究室在内的众多研究机构。
郑伟教授及团队成员胡刚教授与博士生倪文韬与密歇根大学等高校合作,在第16届CASP比赛中斩获多项冠军。在蛋白质结构预测方面,蛋白质单体单结构域预测赛道评估参赛算法在预测蛋白质基本单元结构的能力,郑伟教授团队在这一赛道中课题组排名第二(基于Z-score > -2.0)。此外,在“hard”类别的蛋白质复合物结构预测中,郑伟教授团队凭借显著优势位居第一。该任务因缺少单体和复合物模板而极具挑战性,对参赛团队的算法设计与预测能力提出了更高要求。
与传统的单一构象预测不同,多构象预测类别需要考虑同一分子在不同环境和条件下可能表现出的多种稳定状态或功能状态,因此给各参赛队伍带来不少困难。而郑伟教授团队凭借出色的预测能力,在这一赛道中TM-score指标排名第一。与蛋白质结构预测相比,核酸结构预测领域因实验数据匮乏而难度倍增。在核酸多聚体结构预测方面,郑伟教授带领的团队在服务器组排名第一(基于Z-score > -2.0)。
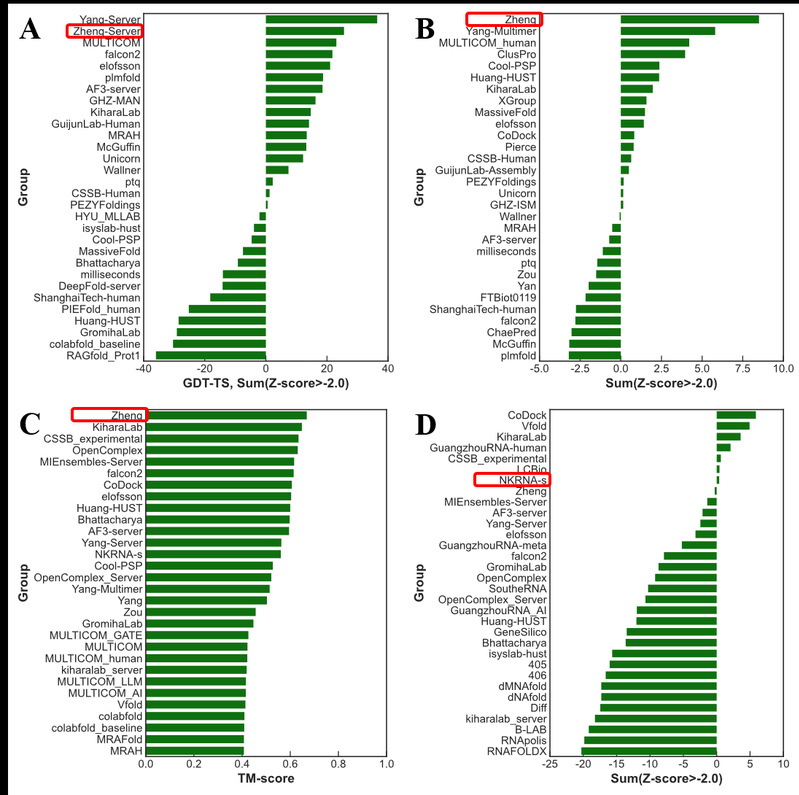
图1. CASP16多项结构预测排名。(A) 蛋白质单体单结构域预测排名(Top 30)。(B) “hard”蛋白质复合物结构预测排名(Top 30)。(C) 大分子多构象预测排名(Top 30)。(D) 核酸多聚体结构预测排名(Top 30)。红色方框为郑伟教授参赛方法。
此外,CASP16首次设立了蛋白质-核酸复合物结构预测赛道,这一赛道要求参赛团队精准捕捉蛋白质与核酸的相互作用模式,并预测其整体空间构象。面对这一全新挑战,郑伟教授团队在众多队伍中脱颖而出,获得第一名。
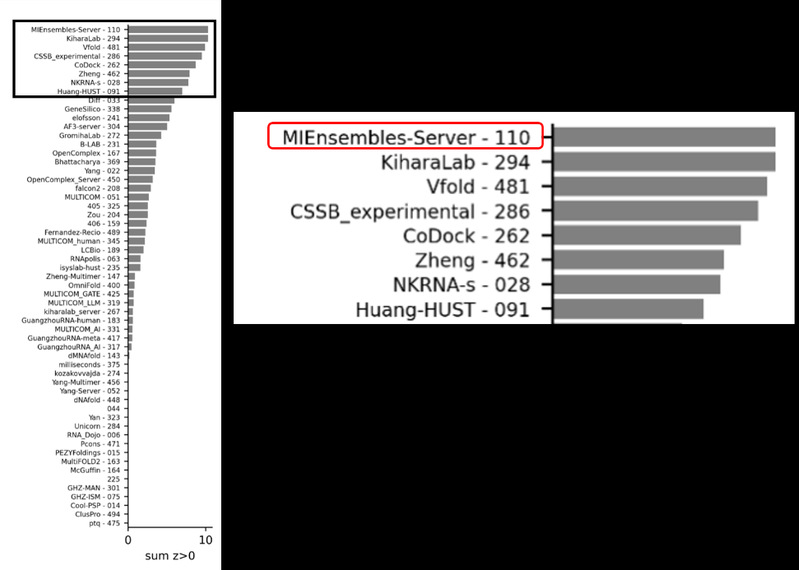
图2. CASP16蛋白质-核酸复合物结构预测排名。红色方框为郑伟教授参赛方法。
模型准确性估计类别要求参赛团队对给定的蛋白质结构模型质量进行评估。郑伟教授团队在复合物整体精度估计(QMODE1)的两个赛道中均跻身前二,其中折叠精度估计排名第一,界面精度估计中排名第二。这两个子任务分别聚焦于复合物整体结构和相互作用区域的准确性评估。此外,团队在单体模型筛选赛道中也表现不俗,排名第三。
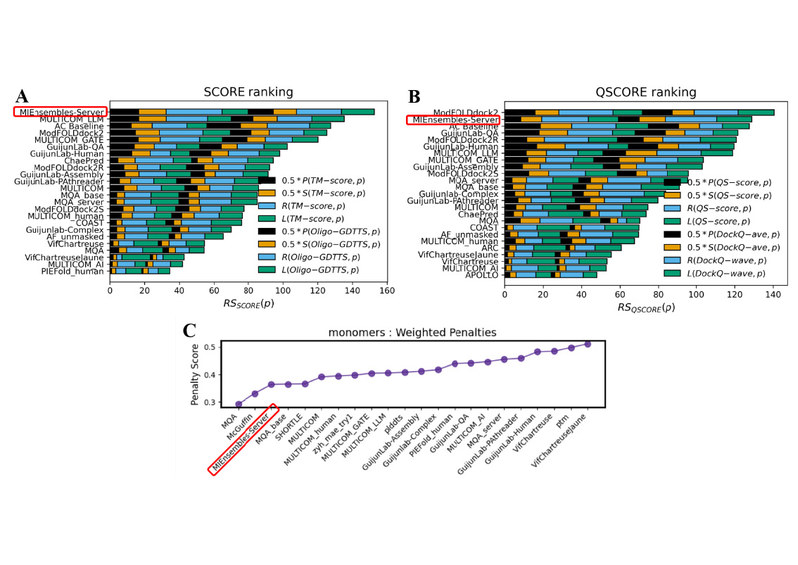
图3. CASP16 准确性估计排名。(A) 复合物整体折叠精度估计(SCORE)排名。(B) 复合物整体界面精度估计(QSCORE)排名。(C) 单体模型筛选排名。红色方框为郑伟教授参赛方法。
近年来,蛋白质结构预测领域持续受到广泛关注,成为生物学和计算生物学的研究热点。2024年诺贝尔化学奖一半授予David Baker,表彰其在计算蛋白质设计方面的贡献,另一半则共同授予Demis Hassabis和John M. Jumper,以表彰他们在蛋白质结构预测方面的贡献。尽管AlphaFold3等先进预测工具也参与了今年的CASP16,郑伟教授团队的预测方法仍显著优于AlphaFold3,展现了团队在这一领域的独特优势和深厚的科研积累。